|
产品名称 |
SK-HEP-1人肝癌细胞 |
|
货号 |
ZQ0030 |
|
产品介绍 |
SK-HEP-1是从患有肝腺癌的52岁男性患者的肝脏中分离的内皮细胞。尽管该细胞系最初被归类为肝癌细胞系,但现有的研究证据表明,它们更准确地应该被视为肝窦内皮细胞的细胞模型,而不是肝癌细胞。因此,SK-HEP-1细胞系在研究肝窦内皮细胞的生物学特性以及在肝病和肝癌研究中的应用中具有重要价值。应用于心血管疾病研究、毒理学和免疫肿瘤学等研究。该细胞染色体在亚三倍体范围内。在裸鼠中,SK-HEP-1细胞能形成与肝癌相一致的大细胞癌。 |
|
种属 |
人 |
|
性别/年龄 |
男/52岁 |
|
组织 |
肝脏/腹水 |
|
疾病 |
腺癌 |
|
生物安全等级 |
BSL -1 |
|
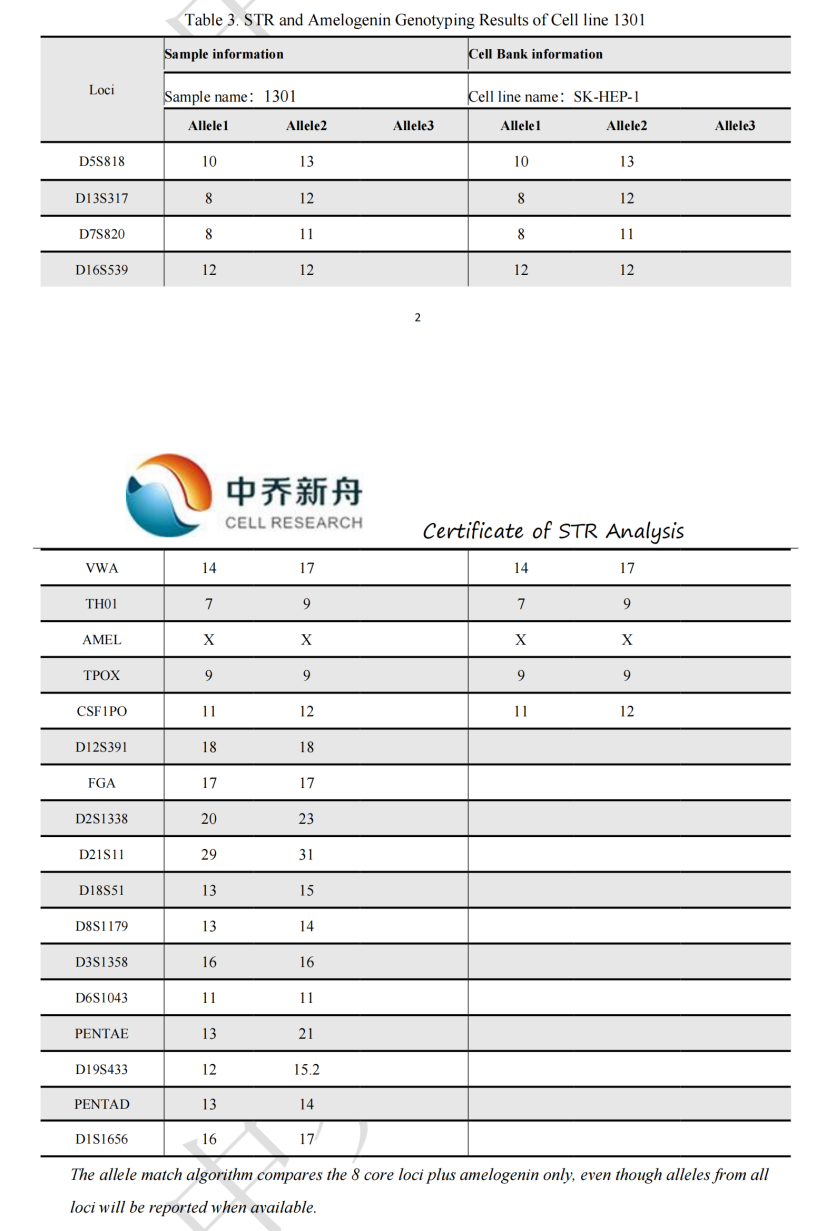
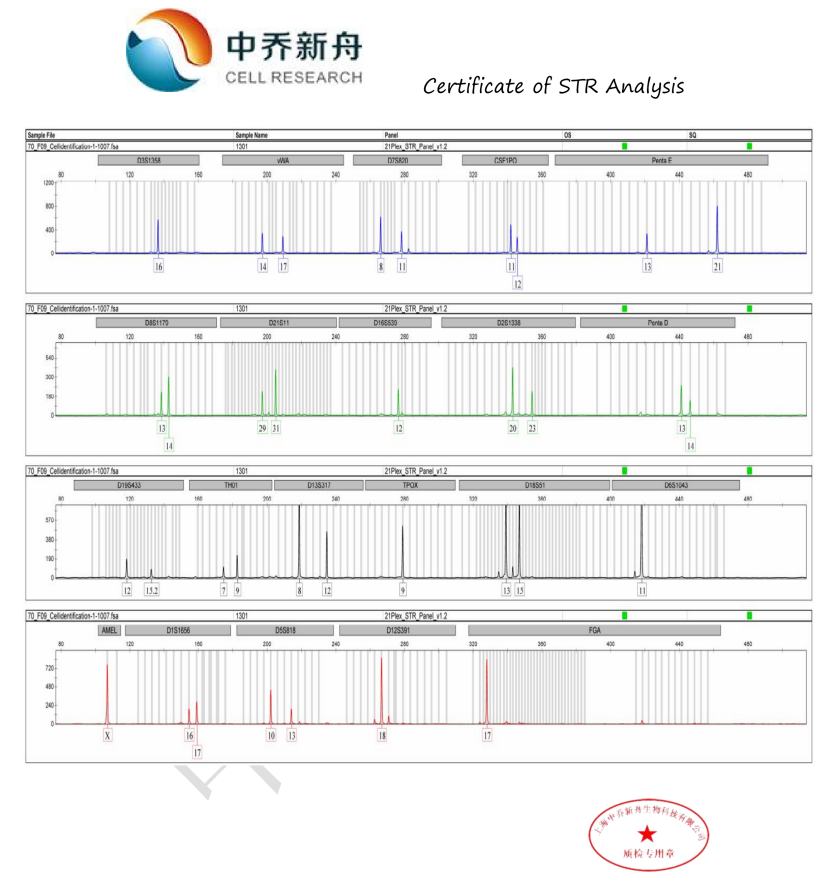
STR位点信息 |
Amelogenin: X
CSF1PO: 11,12
|
|
细胞类型 |
肿瘤细胞 |
|
形态学 |
上皮细胞样 |
|
生长方式 |
贴壁 |
|
倍增时间 |
大约30~46.7小时 |
|
培养基和添加剂 |
|
|
推荐完全培养基货号 |
|
|
培养条件 |
95%空气,5%二氧化碳;37℃ |
|
抗原表达/受体表达 |
*** |
|
基因表达 |
*** |
|
细胞来源 |
ATCC |
|
供应限制 |
仅供科研使用 |
|
货号 |
ZQ0030 |
|
发货规格 |
活细胞:T25培养瓶*1瓶或者1ml 冻存管*2支(细胞量约为1x10^6 Cells/Vial)二选一 |
|
发货形式 |
活细胞:常温运输;冻存管:干冰运输 |
|
储存温度 |
活细胞:培养箱;冻存管:液氮罐 |
|
产地 |
中国 |
|
供应限制 |
仅供科研使用 |
| 货号 | 产品名称 | 规格 | 价格 | 指令 |
 上海中乔新舟生物科技有限公司
上海中乔新舟生物科技有限公司