|
产品名称 |
mo7e人巨细胞白血病细胞 |
|
货号 |
ZQ0934 |
|
产品介绍 |
M-07e细胞系是源自最初的M-07人白血病细胞系的一个亚系,该细胞系是从一名诊断为急性巨核母细胞白血病(AML M7)的6个月大女孩的外周血中建立的。这种特殊的亚系被分离出来,形成一种依赖因子的细胞系,即使在胎儿血清存在的情况下,也需要白细胞介素-3 (IL-3)或粒细胞巨噬细胞集落刺激因子(GM-CSF)来生长。M-07e细胞在多种细胞因子的作用下表现出强劲的增殖反应,包括GM-CSF、干扰素(ifn - α、ifn - β、ifn - γ)、IL-2、IL-3、IL-4、IL-6、IL-15、神经生长因子(NGF)、干细胞因子(SCF)、肿瘤坏死因子- α (tnf - α)和血小板生成素(TPO)。然而,它们对IL-3或GM-CSF的依赖性使它们成为测量这些特定细胞因子生物活性的生物测定中有价值的工具。值得注意的是,M-07e细胞对IL-3和GM-CSF高度敏感,这使得它们非常适合用于检测低水平这些细胞因子的检测。例如,使用M-07e细胞的生物测定可以检测到低至25-50 pg/mL的IL-3或GM-CSF,使其与传统的检测方法(如CFU-GM或CML细胞增殖试验)相当,甚至更敏感。然而,在培养的3-4周内,细胞系有变得不依赖细胞因子的趋势,可能是由于细胞因子不依赖亚群的生长,这表明在使用这些细胞进行长期研究时,需要仔细监测。外显子组和RNA序列数据的可用性进一步增强了M-07e细胞在白血病和造血研究中的实用性。
M-07e细胞也被用于建立GM-CSF和IL-3的定量生物测定,这在临床和研究环境中都是必不可少的。利用该细胞系开发的生物测定法已被证明是方便、可靠和敏感的,使其在评估造血生长因子治疗的药理作用方面特别有用。M-07e细胞对各种细胞因子的详细反应性,结合其充分记录的生长特征,强调了它们在实验血液学中的价值,特别是在与白血病和细胞因子治疗应用相关的研究中。 |
|
种属 |
人 |
|
性别/年龄 |
女/6个月 |
|
组织 |
外周血 |
|
疾病 |
急性巨核细胞白血病 |
|
细胞类型 |
肿瘤细胞 |
|
形态学 |
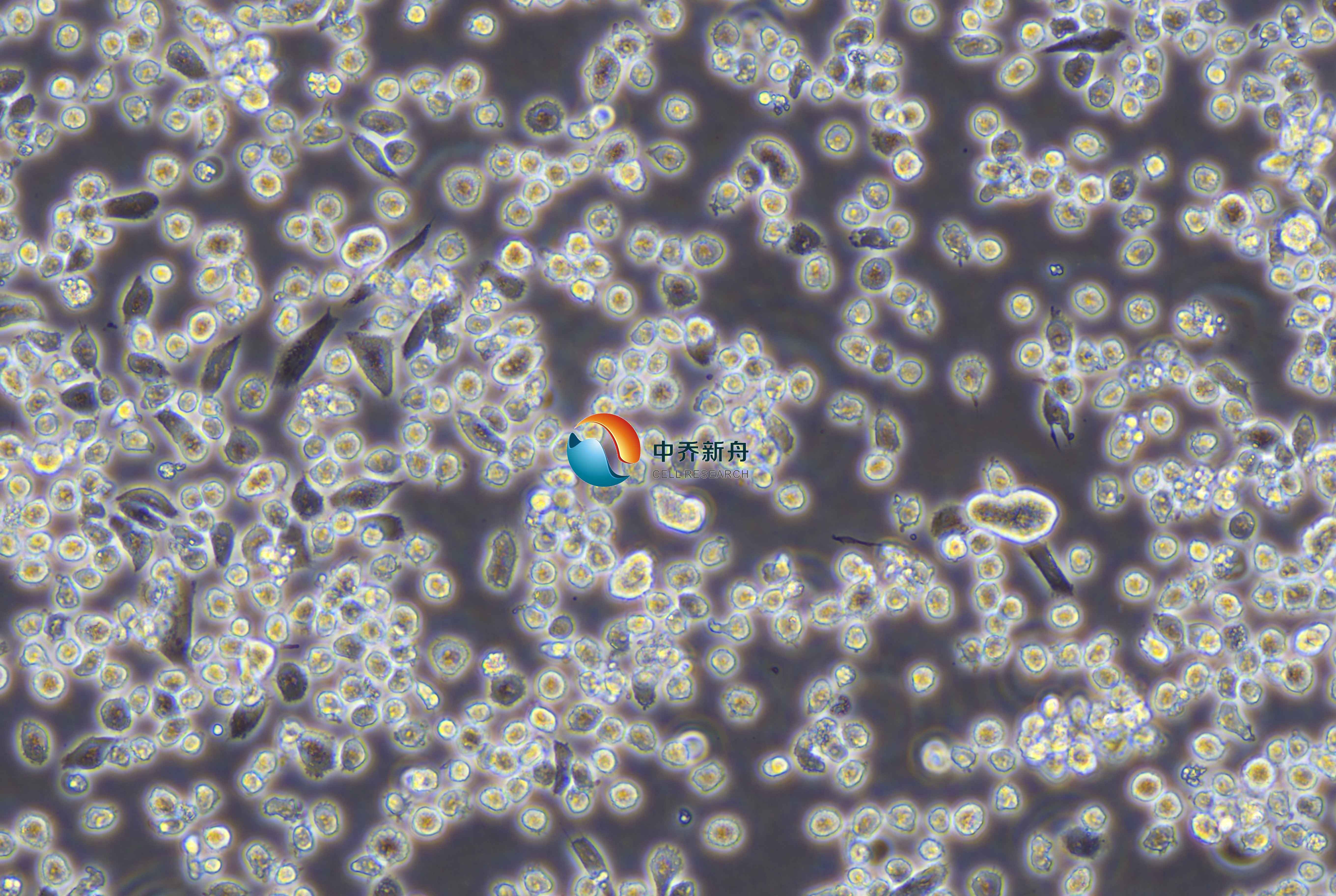
单个圆形细胞悬液中,少数细胞可呈梭形,稍有贴壁 |
|
生长方式 |
半贴半悬 |
|
倍增时间 |
大约34~58小时 |
|
培养基和添加剂 |
RPMI-1640基础培养基(中乔新舟 货号:ZQ-200)+10%FBS(中乔新舟 货号:ZQ0500)+rHuGM-csf 8ng/ml (中乔新舟 货号:CSP023)+1%PS(中乔新舟 货号:CSP006) |
|
推荐完全培养基货号 |
|
|
生物安全等级 |
BSL-1 |
|
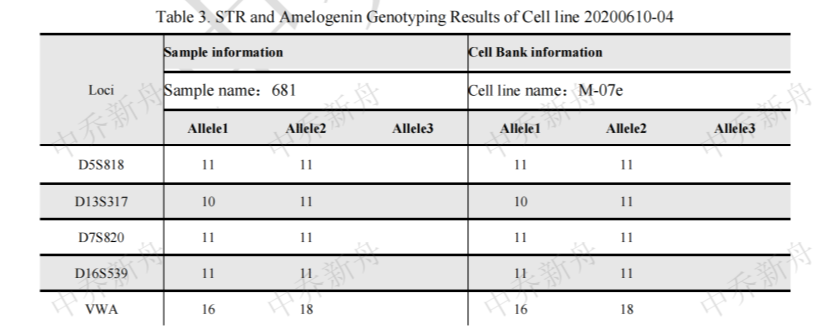
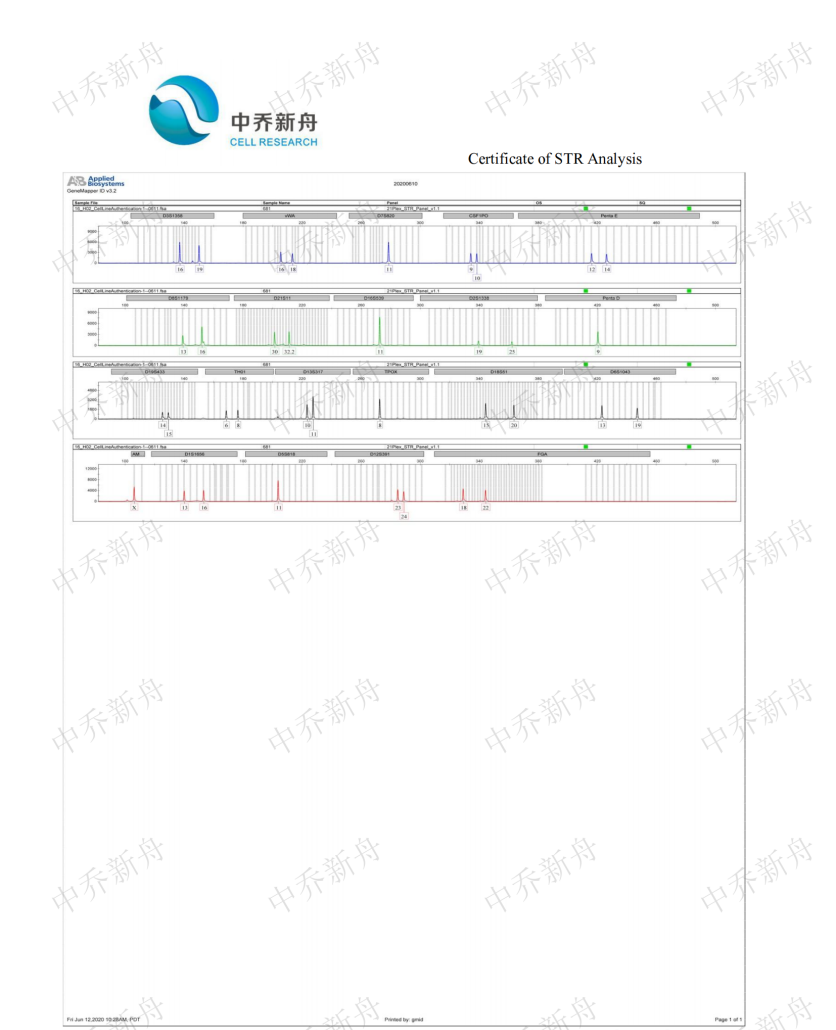
STR位点信息 |
Amelogenin:X |
|
培养条件 |
95%空气,5%二氧化碳;37℃ |
|
抗原表达/受体表达 |
*** |
|
基因表达 |
*** |
|
保藏机构 |
DSMZ; ACC-104 |
|
供应限制 |
仅供科研使用 |
|
货号 |
ZQ0934 |
|
发货规格 |
活细胞:T25培养瓶*1瓶或者1ml 冻存管*2支(细胞量约为1x10^6 Cells/Vial)二选一 |
|
发货形式 |
活细胞:常温运输;冻存管:干冰运输 |
|
储存温度 |
活细胞:培养箱;冻存管:液氮罐 |
|
产地 |
中国 |
|
供应限制 |
仅供科研使用 |
PubMed=2273055; DOI=10.1002/jcp.1041450310
Avanzi G.C., Brizzi M.F., Giannotti J., Brennan A., Yang Y.-C., Pegoraro L., Clark S.C.
M-07e human leukemic factor-dependent cell line provides a rapid and sensitive bioassay for the human cytokines GM-CSF and IL-3.
J. Cell. Physiol. 145:458-464(1990)
PubMed=9096695; DOI=10.1038/sj.leu.2400621
Drexler H.G., Zaborski M., Quentmeier H.
Thrombopoietin supports the continuous growth of cytokine-dependent human leukemia cell lines.
Leukemia 11:541-551(1997)
DOI=10.1016/B978-0-12-221970-2.50457-5
Drexler H.G.
The leukemia-lymphoma cell line factsbook.
(In) ISBN 9780122219702; pp.1-733; Academic Press; London (2001)
PubMed=11414198; DOI=10.1007/s004320000207
Lahm H., Andre S., Hoeflich A., Fischer J.R., Sordat B., Kaltner H., Wolf E., Gabius H.-J.
Comprehensive galectin fingerprinting in a panel of 61 human tumor cell lines by RT-PCR and its implications for diagnostic and therapeutic procedures.
J. Cancer Res. Clin. Oncol. 127:375-386(2001)
PubMed=16408098; DOI=10.1038/sj.leu.2404081
Quentmeier H., MacLeod R.A.F., Zaborski M., Drexler H.G.
JAK2 V617F tyrosine kinase mutation in cell lines derived from myeloproliferative disorders.
Leukemia 20:471-476(2006)
PubMed=22460905; DOI=10.1038/nature11003
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=28109323; DOI=10.1186/s13045-017-0396-0
Masetti R., Bertuccio S.N., Astolfi A., Chiarini F., Lonetti A., Indio V., De Luca M., Bandini J., Serravalle S., Franzoni M., Pigazzi M., Martelli A.M., Basso G., Locatelli F., Pession A.
Hh/Gli antagonist in acute myeloid leukemia with CBFA2T3-GLIS2 fusion gene.
J. Hematol. Oncol. 10:26.1-26.5(2017)
PubMed=30629668; DOI=10.1371/journal.pone.0210404
Uphoff C.C., Pommerenke C., Denkmann S.A., Drexler H.G.
Screening human cell lines for viral infections applying RNA-Seq data analysis.
PLoS ONE 14:E0210404-E0210404(2019)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. III, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
PubMed=31160637; DOI=10.1038/s41598-019-44491-x
Quentmeier H., Pommerenke C., Dirks W.G., Eberth S., Koeppel M., MacLeod R.A.F., Nagel S., Steube K., Uphoff C.C., Drexler H.G.
The LL-100 panel: 100 cell lines for blood cancer studies.
Sci. Rep. 9:8218-8218(2019)

 上海中乔新舟生物科技有限公司
上海中乔新舟生物科技有限公司