配套完培,组合使用,省时省力!
|
产品名称 |
NK-92MI人恶性非霍奇金淋巴瘤患者的自然杀伤细胞 |
|
货号 |
ZQ0435 |
|
产品介绍 |
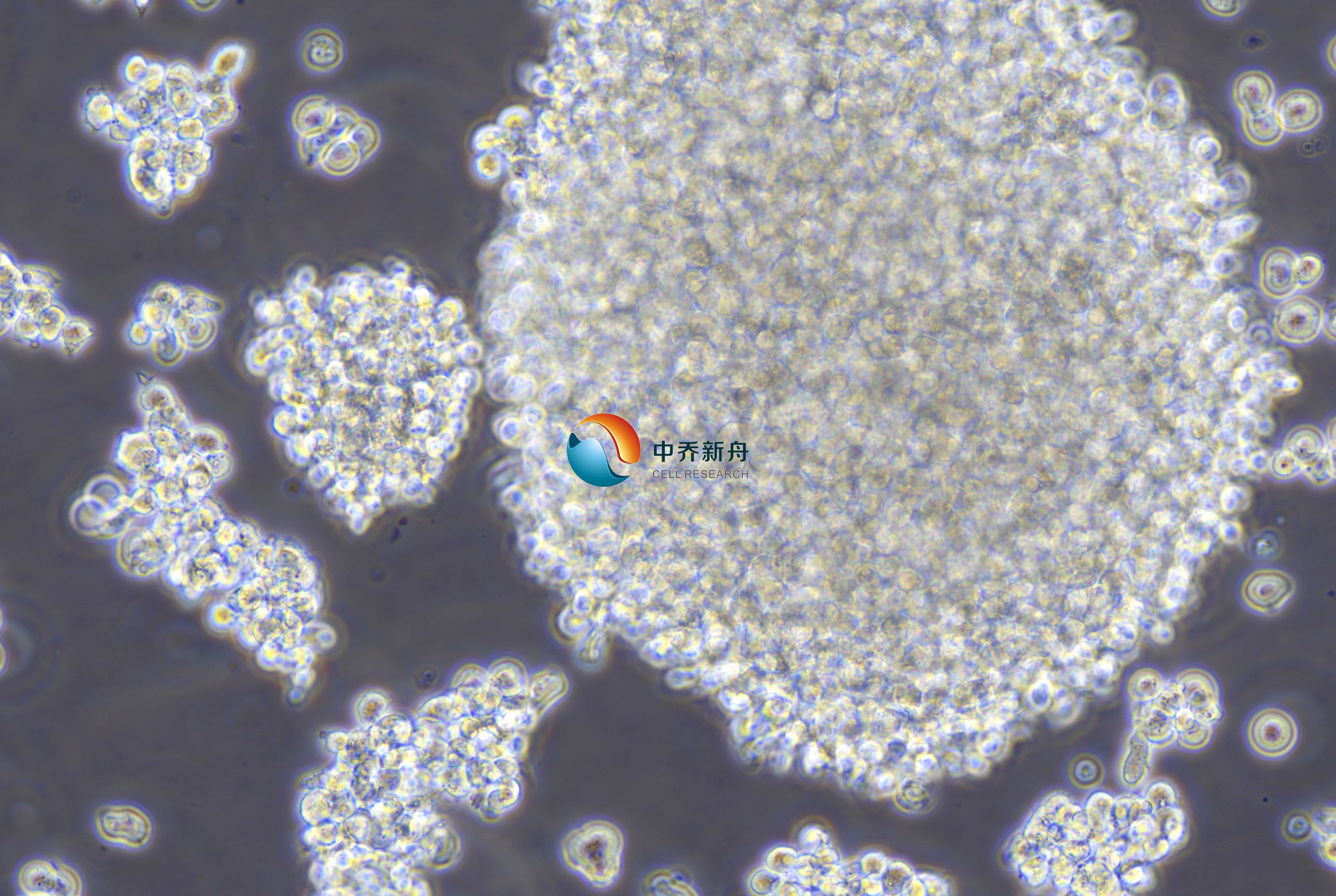
NK-92细胞是从一位患有急进性非霍奇金淋巴瘤的50岁白人男性外周血单核细胞衍生来的一株IL-2依赖型NK细胞株。NK-92MI细胞是转染得到的源自NK-92细胞的IL-2非依赖的NK细胞株。亲本细胞NK-92通过微粒体基因转化法用逆转录病毒MFG-hIL-2载体携带的人IL-2cDNA进行转化。可能由于载体整合到基因组DNA中,转化是稳定的。这株细胞对很多恶性细胞有细胞毒性;铬释放试验显示它能杀死K562细胞和Daudi细胞。NK-92细胞有以下特征:CD2、CD7、CD11a、CD28、CD45、CD54表面标记阳性;CD1、CD3、CD4、CD5、CD8、CD10、CD14、CD16、CD19、CD20、CD23、CD34和HLA-DR表面标记阴性。其亲本IL-2依赖的细胞株NK-92细胞及另一株同样来源于NK-92细胞株的IL-2非依赖的细胞株NK-92CI都可从ATCC得到。NK-92MI细胞和NK-92CI细胞这两个变种都包含、表达并合成hIL-2cDNA。NK-92MI细胞合成的IL-2水平比NK-92CI高,而亲本细胞不合成表达。1998年9月提交到ATCC的培养物污染了支原体,其后代通过BM细胞周期蛋白处理21天消除支原体。处理后6周,用Hoechst染色、PCR和标准培养测试进行支原体检测,结果都呈阴性。
注意事项: |
|
种属 |
人 |
|
性别/年龄 |
男/50岁 |
|
组织 |
外周血 |
|
疾病 |
淋巴瘤;恶性非霍奇金淋巴瘤 |
|
细胞类型 |
自然杀伤细胞;nk细胞 |
|
形态学 |
淋巴样 |
|
生长方式 |
悬浮;多细胞聚集体 |
|
培养基和添加剂 |
MEMα(中乔新舟 货号:ZQ-800)+12.5%热灭活马血清(中乔新舟 货号:ZQH500)+12.5%胎牛血清(中乔新舟 货号:ZQ0500)+1%双抗(中乔新舟 货号:CSP006)+0.2mM肌醇+0.02mM叶酸+0.1mM β-Mer(中乔新舟 货号:CSP005) |
|
推荐完全培养基货号 |
|
|
生物安全等级 |
BSL-2 |
|
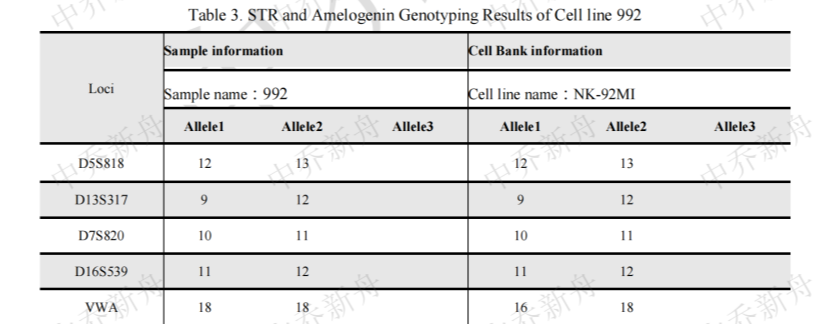
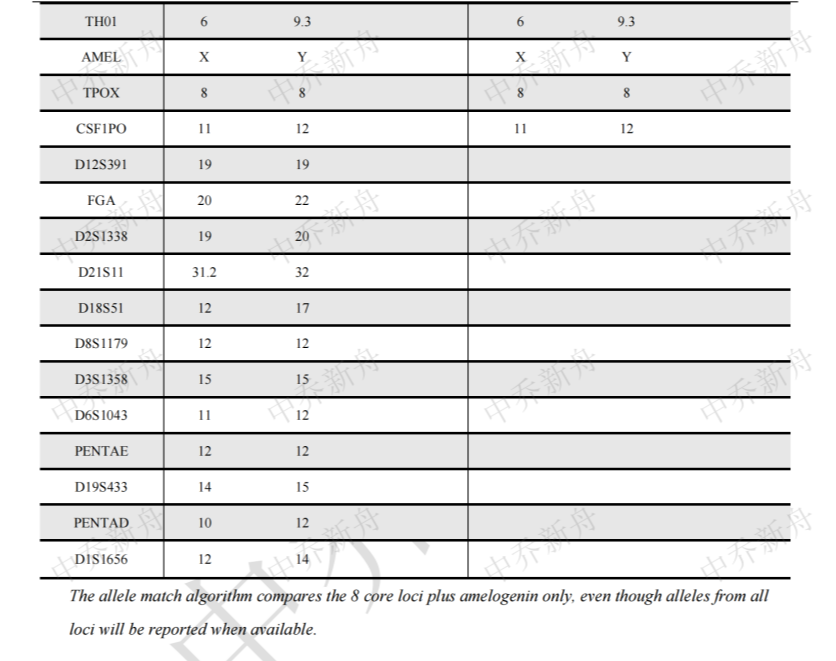
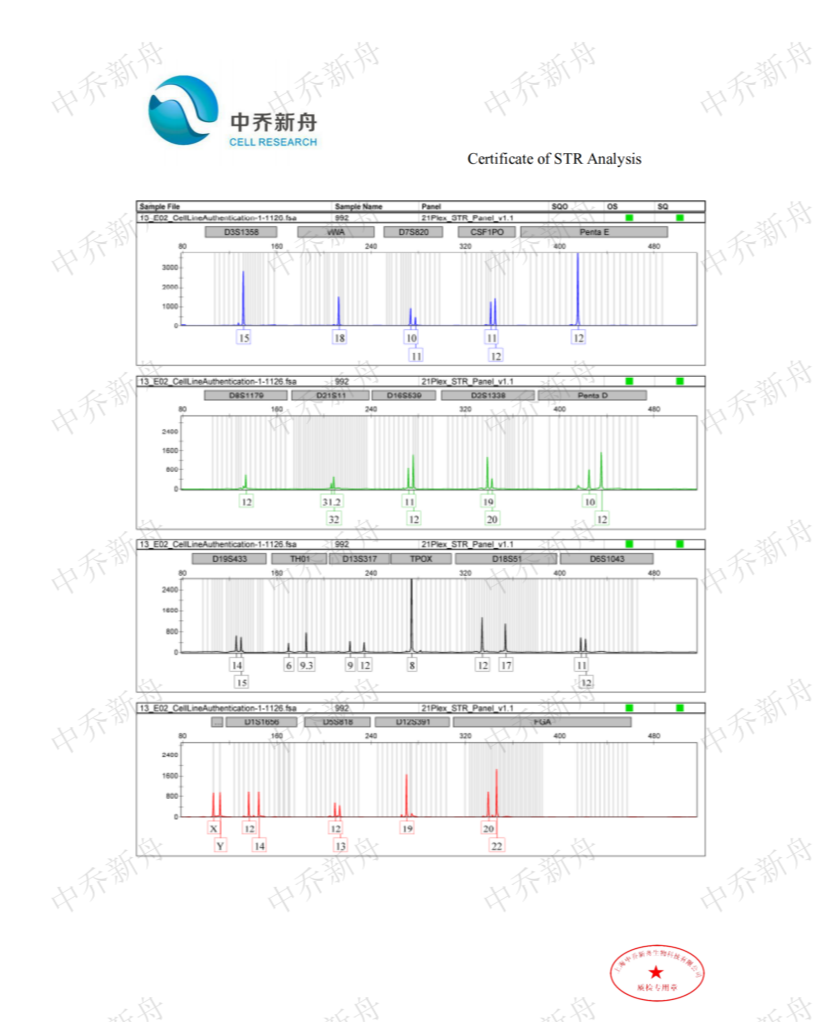
STR位点信息 |
D5S818: 12. 13 D13S317: 9, 12 D7S820: 10, 11 D16S539: 11, 12 vWA: 16, 18 THO1: 6, 9.3 TPOX: 8 CSF1PO: 11, 12 Amelogenin: X, Y |
|
培养条件 |
95%空气,5%二氧化碳;37℃ |
|
抗原表达/受体表达 |
*** |
|
基因表达 |
*** |
|
保藏机构 |
ATCC; CRL-2408 |
|
供应限制 |
仅供科研使用 |
|
货号 |
ZQ0435 |
|
发货规格 |
活细胞:T25培养瓶*1瓶或者1ml 冻存管*2支(细胞量约为1x10^6 Cells/Vial)二选一 |
|
发货形式 |
活细胞:常温运输;冻存管:干冰运输 |
|
储存温度 |
活细胞:培养箱;冻存管:液氮罐 |
|
产地 |
中国 |
|
供应限制 |
仅供科研使用 |
PubMed=10365666; DOI=10.1089/10430349950018030
Tam Y.K., Maki G., Miyagawa B., Hennemann B., Tonn T., Klingemann H.-G.
Characterization of genetically altered, interleukin 2-independent natural killer cell lines suitable for adoptive cellular immunotherapy.
Hum. Gene Ther. 10:1359-1373(1999)
Patent=US8034332
Klingemann H.-G.
Interleukin-secreting natural killer cell lines and methods of use.
Patent number US8034332, 11-Oct-2011
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Mironenko T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=35839778; DOI=10.1016/j.ccell.2022.06.010
Goncalves E., Poulos R.C., Cai Z.-X., Barthorpe S., Manda S.S., Lucas N., Beck A., Bucio-Noble D., Dausmann M., Hall C., Hecker M., Koh J., Lightfoot H., Mahboob S., Mali I., Morris J., Richardson L., Seneviratne A.J., Shepherd R., Sykes E., Thomas F., Valentini S., Williams S.G., Wu Y.-X., Xavier D., MacKenzie K.L., Hains P.G., Tully B., Robinson P.J., Zhong Q., Garnett M.J., Reddel R.R.
Pan-cancer proteomic map of 949 human cell lines.
Cancer Cell 40:835-849.e8(2022)
 上海中乔新舟生物科技有限公司
上海中乔新舟生物科技有限公司