|
产品名称 |
HuH-6人肝母细胞瘤细胞 |
|
货号 |
ZQ0246 |
|
产品介绍 |
HuH-6 细胞是人类肝癌 6 细胞的简称,是一种永久性细胞系,由 1982 年从一名 57 岁的男性体内获得的肝癌组织建立而成。
注意事项: |
|
种属 |
人 |
|
性别/年龄 |
男/1岁 |
|
组织 |
肝 |
|
疾病 |
肝母细胞瘤 |
|
细胞类型 |
肿瘤细胞 |
|
形态学 |
上皮细胞 |
|
生长方式 |
贴壁 |
|
倍增时间 |
大约3-5天 |
|
培养基和添加剂 |
DMEM高糖(中乔新舟 货号:ZQ-100)+10%胎牛血清(中乔新舟 货号:ZQ0500)+1%双抗(中乔新舟 货号:CSP006) |
|
推荐完全培养基货号 |
|
|
生物安全等级 |
BSL-1 |
|
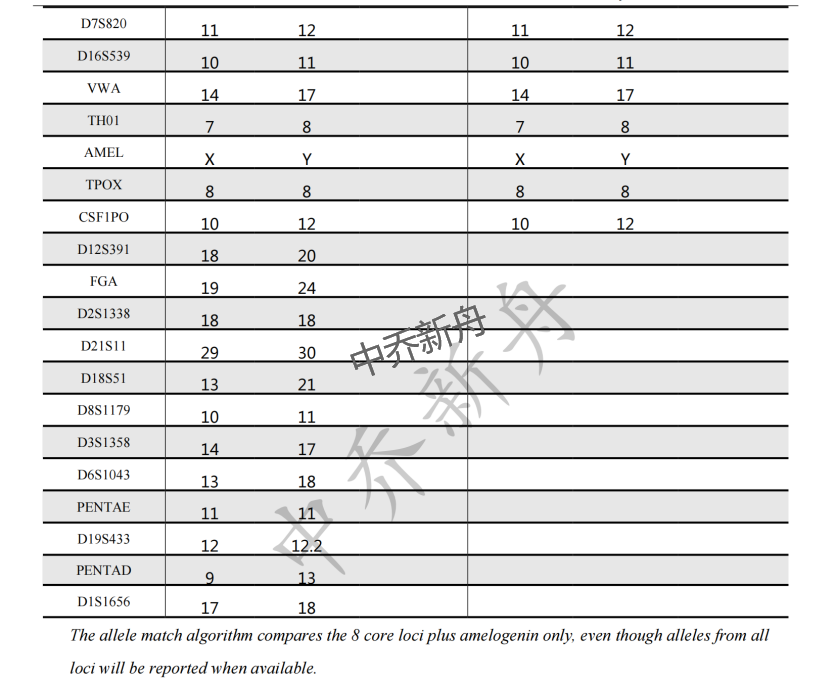
STR位点信息 |
Amelogenin:X,Y; CSF1PO:10,12; D13S317:8,12; D16S539:10,11; D18S51:13,21; D19S433:12,12.2; D21S11:29,30; D2S1338:18; D3S1358:14,17; D5S818:8,11; D7S820:11,12; D8S1179:10,11; FGA:19,24; TH01:7,8; TPOX:8; vWA:14,17 |
|
培养条件 |
95%空气,5%二氧化碳;37℃ |
|
抗原表达/受体表达 |
*** |
|
基因表达 |
*** |
|
保藏机构 |
RCB |
|
供应限制 |
仅供科研使用 |
|
货号 |
ZM0246 |
|
发货规格 |
活细胞:T25培养瓶*1瓶或者1ml 冻存管*2支(细胞量约1x10^6 Cells/Vial)二选一 |
|
发货形式 |
活细胞:常温运输;冻存管:干冰运输 |
|
储存温度 |
活细胞:培养箱;冻存管:液氮罐 |
|
产地 |
中国 |
|
供应限制 |
仅供科研使用 |
PubMed=57894
Doi I.
Establishment of a cell line and its clonal sublines from a patient with hepatoblastoma.
Gann 67:1-10(1976)
DOI=10.11418/jtca1981.12.3_221
Namba M., Nakabayashi H., Doi I., Sato J., Miyazaki M.
Cellular characteristics and utilization of human hepatoma cell lines which were established in our laboratory and distributed by Japanese Cancer Research Resources Bank.
Tissue Cult. Res. Commun. 12:221-227(1993)
PubMed=8389256; DOI=10.1093/carcin/14.5.987
Hsu I.C., Tokiwa T., Bennett W., Metcalf R.A., Welsh J.A., Sun T., Harris C.C.
p53 gene mutation and integrated hepatitis B viral DNA sequences in human liver cancer cell lines.
Carcinogenesis 14:987-992(1993)
DOI=10.11418/jtca1981.16.3_173
Mihara K., Miyazaki M., Fushimi K., Tsuji T., Inoue Y., Fukaya K.-i., Ohashi R., Namba M.
The p53 gene status and other cellular characteristics of human cell lines maintained in our laboratory.
Tissue Cult. Res. Commun. 16:173-178(1997)
PubMed=9359923; DOI=10.18926/AMO/30789
Mihara K., Miyazaki M., Kondo T., Fushimi K., Tsuji T., Inoue Y., Fukaya K.-i., Ishioka C., Namba M.
Yeast functional assay of the p53 gene status in human cell lines maintained in our laboratory.
Acta Med. Okayama 51:261-265(1997)
PubMed=9671767; DOI=10.1073/pnas.95.15.8847
de la Coste A., Romagnolo B., Billuart P., Renard C.-A., Buendia M.-A., Soubrane O., Fabre M., Chelly J., Beldjord C., Kahn A., Perret C.
Somatic mutations of the beta-catenin gene are frequent in mouse and human hepatocellular carcinomas.
Proc. Natl. Acad. Sci. U.S.A. 95:8847-8851(1998)
PubMed=15767549; DOI=10.1158/1535-7163.MCT-04-0234
Nakatsu N., Yoshida Y., Yamazaki K., Nakamura T., Dan S., Fukui Y., Yamori T.
Chemosensitivity profile of cancer cell lines and identification of genes determining chemosensitivity by an integrated bioinformatical approach using cDNA arrays.
Mol. Cancer Ther. 4:399-412(2005)
PubMed=20937217; DOI=10.1170/149
Di Masi A., Viganotti M., Antoccia A., Magrelli A., Salvatore M., Azzalin G., Tosto F., Lorenzetti S., Maranghi F., Mantovani A., Macino G., Tanzarella C., Taruscio D.
Characterization of HuH6, Hep3B, HepG2 and HLE liver cancer cell lines by WNT/beta-catenin pathway, microRNA expression and protein expression profile.
Cell. Mol. Biol. 56:OL1299-OL1317(2010)
PubMed=22460905; DOI=10.1038/nature11003
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=23505090; DOI=10.1002/hep.26402
Wang K., Lim H.Y., Shi S., Lee J., Deng S.-B., Xie T., Zhu Z., Wang Y.-L., Pocalyko D., Yang W.J., Rejto P.A., Mao M., Park C.-K., Xu J.-C.
Genomic landscape of copy number aberrations enables the identification of oncogenic drivers in hepatocellular carcinoma.
Hepatology 58:706-717(2013)
PubMed=23887712; DOI=10.1038/ncomms3218
Nault J.-C., Mallet M., Pilati C., Calderaro J., Bioulac-Sage P., Laurent C., Laurent A., Cherqui D., Balabaud C., Zucman-Rossi J.
High frequency of telomerase reverse-transcriptase promoter somatic mutations in hepatocellular carcinoma and preneoplastic lesions.
Nat. Commun. 4:2218.1-2218.7(2013)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31063779; DOI=10.1053/j.gastro.2019.05.001
Caruso S., Calatayud A.-L., Pilet J., La Bella T., Rekik S., Imbeaud S., Letouze E., Meunier L., Bayard Q., Rohr-Udilova N., Peneau C., Grasl-Kraupp B., de Koning L., Ouine B., Bioulac-Sage P., Couchy G., Calderaro J., Nault J.-C., Zucman-Rossi J., Rebouissou S.
Analysis of liver cancer cell lines identifies agents with likely efficacy against hepatocellular carcinoma and markers of response.
Gastroenterology 157:760-776(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. III, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
PubMed=31978347; DOI=10.1016/j.cell.2019.12.023
Nusinow D.P., Szpyt J., Ghandi M., Rose C.M., McDonald E.R. III, Kalocsay M., Jane-Valbuena J., Gelfand E.T., Schweppe D.K., Jedrychowski M.P., Golji J., Porter D.A., Rejtar T., Wang Y.K., Kryukov G.V., Stegmeier F., Erickson B.K., Garraway L.A., Sellers W.R., Gygi S.P.
Quantitative proteomics of the Cancer Cell Line Encyclopedia.
Cell 180:387-402.e16(2020)
PubMed=32899426; DOI=10.3390/cancers12092510
Scherer D., Davila-Lopez M., Goeppert B., Abrahamsson S., Gonzalez Silos R., Nova I., Marcelain K., Roa J.C., Ibberson D., Umu S.U., Rounge T.B., Roessler S., Lorenzo-Bermejo J.
RNA sequencing of hepatobiliary cancer cell lines: data and applications to mutational and transcriptomic profiling.
Cancers (Basel) 12:2510.1-2510.14(2020)


| 产品名称 | 价格 | 指令 |
| MHCC97-L人肝癌细胞(低转)(STR鉴定)[细胞+500ml专培套餐促销] | ¥980 | 购物车 》 |
| Bel-7404人肝癌细胞(hela污染) | ¥1400.00 | 购物车 》 |
| Hep 3B2.1-7人肝癌细胞(STR鉴定)[细胞+500ml专培套餐促销] | ¥980.00 | 购物车 》 |
| huh-7人肝癌细胞(STR鉴定) | ¥1600.00 | 购物车 》 |
| A172人胶质母细胞瘤细胞(STR鉴定)[细胞+500ml专培套餐促销] | ¥980.00 | 购物车 》 |
| SH-SY5Y人神经母细胞瘤细胞(STR鉴定) | ¥1350.00 | 购物车 》 |
| SK-N-AS人神经母细胞瘤细胞(STR鉴定)[细胞+500ml专培套餐促销] | ¥980.00 | 购物车 》 |
| U-87 MG人脑星形胶质母细胞瘤(STR鉴定)[细胞+500ml专培套餐促销] | ¥980 | 购物车 》 |
| MOLT-4人急性淋巴母细胞白血病细胞(STR鉴定)[细胞+500ml专培套餐促销] | ¥980.00 | 购物车 》 |
| 支原体检测试剂盒(买二送一) | ¥800.00 | 购物车 》 |
| 支原体清除试剂盒 | ¥600.00 | 购物车 》 |
| 国产转染试剂 | ¥800.00 | 购物车 》 |
| 国产优级胎牛血清 | ¥1800.00 | 购物车 》 |
| DMEM高糖基础培养基 | ¥56.00 | 购物车 》 |
| DMEM高糖完全培养基(10%FBS) | ¥350.00 | 购物车 》 |
 上海中乔新舟生物科技有限公司
上海中乔新舟生物科技有限公司